Proteína
Fundo para as escolas Wikipédia
Crianças SOS têm produzido uma seleção de artigos da Wikipédia para escolas desde 2005. Todas as crianças disponíveis para apadrinhamento de crianças de Crianças SOS são cuidadas em uma casa de família pela caridade. Leia mais ...
Proteínas (pron .: / /; também conhecido como polipéptidos) são compostos orgânicos feitos de aminoácidos dispostos numa cadeia linear e dobrados numa globular ou forma fibrosa. Os aminoácidos numa polímero são unidas em conjunto pela ligações peptídicas entre os carboxilo e amino grupos de aminoácido adjacente resíduos. O sequência de aminoácidos de uma proteína é definido pela sequência de uma do gene, que é codificado no código genético . Em geral, o código genético especifica 20 aminoácidos padrão; no entanto, em certos organismos o código genético podem incluir -selenocisteína e em certa archaea- pyrrolysine. Pouco tempo depois, ou mesmo, durante a síntese, os resíduos de uma proteína são muitas vezes quimicamente modificados pela modificação pós-translacional, que altera as propriedades físicas e químicas, dobragem, estabilidade, actividade, e, finalmente, a função das proteínas. As proteínas também podem trabalhar em conjunto para alcançar uma determinada função, e muitas vezes eles associam para formar estável complexos.
Uma das características mais distintivas de polipeptídeos é a sua capacidade de dobrar em um estado globular, ou "estrutura". A medida em que as proteínas se dobram numa estrutura definida varia amplamente. Algumas proteínas dobra em uma estrutura altamente rígida com pequenas flutuações e, portanto, são considerados como estrutura única. Outras proteínas passam por grandes rearranjos de um conformação para outro. Esta mudança conformacional é muitas vezes associada a uma sinalização evento. Assim, a estrutura de uma proteína serve como um meio através do qual a regulação ou a função de uma proteína ou da actividade de uma enzima. Nem todas as proteínas que requerem um processo de dobramento para funcionar, como alguma função em um estado desdobrado.
À semelhança de outros biológica tais como macromoléculas e polissacáridos ácidos nucleicos, as proteínas são componentes essenciais de organismos e participar em praticamente todos os processos dentro de células . Muitas proteínas são enzimas que catalisam reacções bioquímicas e são vitais para metabolismo. As proteínas também tem funções estruturais ou mecânicas, tais como a actina ea miosina de músculo e as proteínas no citoesqueleto, que formam um sistema de andaimes que mantém a forma da célula. Outras proteínas são importantes em sinalização celular, resposta imune , adesão celular, e a ciclo celular. As proteínas também são necessárias na dieta dos animais, uma vez que os animais não podem sintetizar todos os aminoácidos de que necessitam e devem obter aminoácidos essenciais dos alimentos. Através do processo de digestão, animais quebrar as proteínas ingeridas em aminoácidos livres que são em seguida utilizadas no metabolismo.
As proteínas foram descritas pela primeira vez pela Químico holandês Gerhardus Johannes Mulder e nomeado pelo químico sueco Jöns Jakob Berzelius em 1838. cientistas nutricional precoce, como o alemão Carl von Voit acreditava que a proteína foi o nutriente mais importante para a manutenção da estrutura do corpo, pois acreditava-se geralmente que "carne faz com que a carne." O papel central de proteínas como enzimas em organismos vivos foi, porém, não totalmente apreciado até 1926, quando James B. Sumner mostrou que a enzima urease era na verdade uma proteína. A primeira proteína seja sequenciado foi de insulina , por Frederick Sanger , que ganhou o Prêmio Nobel por essa conquista, em 1958. O primeiro estruturas de proteína a ser resolvido eram e hemoglobina mioglobina, por Max Perutz e Sir John Cowdery Kendrew, respectivamente, em 1958. As estruturas tridimensionais de ambas as proteínas foram determinados por primeiro Análise de difracção de raios-X; Perutz e Kendrew compartilhada de 1962 Prêmio Nobel de Química por essas descobertas. As proteínas podem ser purificado a partir de outros componentes celulares usando uma variedade de técnicas, tais como ultracentrifugação, precipitação, electroforese e cromatografia ; o advento de engenharia genética fez possível um certo número de métodos para facilitar a purificação. Os métodos geralmente utilizados para estudar a estrutura e função da proteína incluem imuno-histoquímica, A mutagénese dirigida ao local, ressonância magnética nuclear e espectrometria de massa .
Bioquímica
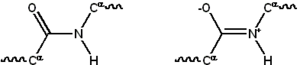
A maioria das proteínas são lineares polímeros construído a partir de séries de até 20 L diferente -α- aminoácidos . Todos os aminoácidos possuem características estruturais comuns, incluindo um α-carbono em que um amino , um grupo carboxilo grupo, e um variável cadeia lateral está ligado . Somente prolina difere dessa estrutura de base, uma vez que contém um anel incomum para o grupo amina N-fim, o que obriga a unidade amida CO-NH numa conformação fixada. As cadeias laterais dos aminoácidos padrão, descritos na lista dos aminoácidos padrão, têm uma grande variedade de estruturas químicas e as propriedades; é o efeito combinado de todas as cadeias laterais de aminoácidos de uma proteína que, em última análise determina a sua estrutura tridimensional e a sua reactividade química.
 | 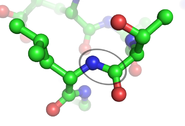 | |
Estrutura química da ligação peptídica (esquerda) e uma ligação peptídica entre e leucina treonina (à direita) | ||
Os aminoácidos de uma cadeia de polipéptido estão ligados por ligações peptídicas. Uma vez ligados na cadeia de proteína, um aminoácido individual é chamado um resíduo, e a série ligada, de azoto, átomos de oxigénio e de carbono são conhecidos como a cadeia principal ou esqueleto da proteína. A ligação peptídica tem dois formas de ressonância que contribuem alguns carácter da ligação dupla e inibir a rotação em torno do seu eixo, de modo que os átomos de carbono alfa são mais ou menos coplanares. Os outros dois ângulos diedros da ligação peptídica determinar a forma local, assumido pelo esqueleto da proteína. O final da proteína com um grupo carboxilo livre é conhecido como o Terminal C ou terminal carboxilo, enquanto que a extremidade com um grupo amino livre é conhecido como o Terminal N ou no terminal amino.
A proteína palavras, polipéptido, e péptido são um pouco ambígua e podem sobrepor-se em sentido. A proteína é geralmente utilizado para se referir à molécula biológica completa num estábulo conformação, enquanto péptido é geralmente reservada para uma curta oligómeros de aminoácidos que faltam muitas vezes uma estrutura tridimensional estável. No entanto, o limite entre os dois não está bem definida e, geralmente, encontra-se perto de 20-30 resíduos. Polipéptido pode referir-se a qualquer cadeia linear única de aminoácidos, independentemente do comprimento normalmente, mas muitas vezes implica a ausência de um definida conformação.
Síntese

As proteínas são montados a partir de aminoácidos utilizando informações codificadas em genes. Cada proteína tem a sua própria sequência de aminoácidos única que é especificada pela sequência de nucleótidos do gene que codifica esta proteína. O código genético é um conjunto de conjuntos de três nucleótidos chamados codões e cada combinação de três nucleótidos designa um aminoácido, por exemplo agosto ( adenine- uracil- guanina) é o código para metionina. Porque o ADN contém quatro nucleótidos, o número total de codões possíveis é 64; Assim, existe alguma redundância no código genético, com alguns aminoácidos especificados por mais de um codão. Os genes codificados no ADN são primeiro transcrita em pré- RNA mensageiro (mRNA) por proteínas tais como a Polimerase de ARN. A maioria dos organismos, em seguida, processar a pré-mRNA (também conhecido como um transcrito primário), utilizando diversas formas de modificação pós-transcricional para formar o mRNA maduro, o qual é então utilizada como um molde para a síntese de proteínas pela ribossomo. Em procariotas o ARNm pode ser utilizada logo que é produzida, ou ser ligado por um ribossoma após ter afastado da nucleoid. Em contraste, eucariotas fazer ARNm no núcleo da célula e, em seguida translocam lo em toda a membrana nuclear para o citoplasma, onde a síntese de proteínas, em seguida, tem lugar. A taxa de síntese de proteínas é mais elevada do que em procariotas e eucariotas pode atingir até 20 aminoácidos por segundo.
O processo de síntese de uma proteína a partir de um molde de ARNm é conhecida como tradução. O mRNA é carregado no ribossoma e é lido três nucleótidos de cada vez por cada codão correspondente à sua base emparelhamento anticodão localizado numa transferir molécula de ARN, o que acarreta o aminoácido correspondente ao codão ele reconhece. A enzima sintetase "encargos" moléculas aminoacil tRNA do tRNA com os aminoácidos corretos. O polipéptido em crescimento é muitas vezes denominado na cadeia nascente. As proteínas são sempre biossintetizado a partir de N-terminal de C-terminal.
O tamanho de uma proteína sintetizada pode ser medida pelo número de aminoácidos que contém pelo seu total e massa molecular , o qual é normalmente reportado em unidades de daltons (sinónimo unidades de massa atómica), ou o derivado de unidade quilodalton (kDa). Levedura proteínas são, em média, ácidos 466 aminoácidos de comprimento e 53 kDa em massa. Os maiores proteínas conhecidas são o titins, um componente do músculo sarcómero, com uma massa molecular de quase 3000 kDa e um comprimento total de cerca de 27.000 aminoácidos.
A síntese química
Proteínas curtas também podem ser sintetizados quimicamente por uma família de métodos conhecidos como a síntese de péptidos, que se baseiam técnicas de síntese orgânica, tais como a ligação química para a produção de péptidos com elevado rendimento. A síntese química permite a introdução de aminoácidos não naturais em cadeias de polipeptídeos, tais como a ligação de sondas fluorescentes para cadeias laterais de aminoácidos. Estes métodos são úteis no laboratório bioquímica e biologia celular, embora geralmente não para aplicações comerciais. A síntese química é ineficiente para polipéptidos mais longos do que cerca de 300 aminoácidos, e as proteínas sintetizadas não pode facilmente assumir a sua nativa estrutura terciária. A maioria dos métodos de síntese química proceder de C-terminal para N-terminal, do lado oposto a reacção biológica.
Estrutura
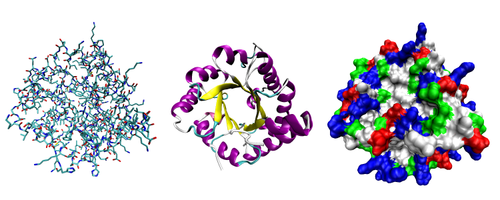
A maioria das proteínas dobrar em estruturas 3 dimensões originais. A forma na qual uma proteína naturalmente dobras é conhecida como o seu conformação nativa. Embora muitas proteínas possam dobrar sem ajuda, simplesmente através das propriedades químicas dos seus aminoácidos, enquanto outros requerem o auxílio de molecular chaperones para dobrar em seus estados de origem. Os bioquímicos muitas vezes referem-se a quatro aspectos distintos da estrutura de uma proteína:
- Estrutura primária: a sequência de aminoácidos.
- Estrutura secundária: que se repete regularmente estruturas locais estabilizados por ligações de hidrogênio. Os exemplos mais comuns são o hélice alfa, folha beta e gira. Como as estruturas secundárias são locais, muitas regiões diferentes da estrutura secundária pode estar presente na mesma molécula de proteína.
- Estrutura terciária: a forma geral de uma única molécula de proteína; a relação espacial das estruturas secundárias para um outro. Estrutura terciária é geralmente estabilizados por interacções não-locais, mais geralmente a formação de um núcleo hidrofóbico, mas também através pontes salinas, ligações de hidrogênio, ligações dissulfureto, e ainda modificações pós-traducionais. O termo "estrutura terciária" é frequentemente utilizado como sinônimo da dobra prazo. A estrutura terciária é o que controla a função básica da proteína.
- A estrutura quaternária: a estrutura formada por várias moléculas de proteína (cadeias polipeptidicas), geralmente chamada subunidades proteicas, neste contexto, que funcionam como uma única complexo de proteína.
As proteínas não são inteiramente moléculas rígidas. Em adição a estes níveis de estrutura, as proteínas podem ser deslocados entre várias estruturas relacionadas, enquanto que desempenhar as suas funções. No contexto destes rearranjos funcionais, estas estruturas terciárias ou quaternárias são geralmente referidos como " conformações ", e transições entre eles são chamados alterações conformacionais. Tais alterações são frequentemente induzidas pela ligação de um molécula de substrato para uma enzima de local activo, ou a região física da proteína que participa na catálise química. Em proteínas de solução também sofrer variação na estrutura por meio de vibração térmica e a colisão com outras moléculas.
As proteínas podem ser informalmente divididas em três classes principais, as quais se correlacionam com as estruturas terciárias típicos: proteínas globulares, proteínas fibrosas, e proteínas da membrana. Quase todas as proteínas globulares são solúveis e muitos são enzimas. Proteínas fibrosas são muitas vezes estrutural, tais como colagénio, o principal componente do tecido conjuntivo, ou queratina, o componente proteína do cabelo e das unhas. Proteínas da membrana muitas vezes servem como receptores ou fornecer canais para polar ou moléculas carregadas para passar através da membrana celular.
Um caso especial de ligações de hidrogênio intramoleculares dentro de proteínas, mal protegido do ataque de água e, consequentemente, promover a sua própria desidratação, são chamados dehydrons.
Determinação de estrutura
Descobrindo a estrutura terciária de uma proteína, ou a estrutura quaternária de seus complexos, pode fornecer pistas importantes sobre como a proteína desempenha a sua função. Métodos experimentais comuns de determinação da estrutura incluir Cristalografia de raios X e Espectroscopia de RMN, ambos os quais podem produzir informações em atômica resolução. No entanto, experiências de RMN são capazes de fornecer informação a partir da qual um subconjunto das distâncias entre pares de átomos pode ser estimado, e as conformações finais possíveis para uma proteína são determinados através da resolução de um problema geometria de distância. Interferometria dupla polarização é um método analítico quantitativo para medir a proteína global conformação e alterações conformacionais devido a interacções ou outro estímulo. Dicroísmo circular é uma outra técnica de laboratório para determinar beta composição folha / helicoidal interna de proteínas. Microscopia cryoelectron é usado para produzir informações estruturais de baixa resolução sobre grandes complexos de proteínas, incluindo montados vírus ; uma variante conhecida como cristalografia de electrões pode também produzir informação de alta-resolução, em alguns casos, especialmente para cristais bidimensionais de proteínas de membrana. Estruturas resolvidas são normalmente depositados no Protein Data Bank (PDB), um recurso disponível livremente a partir do qual os dados estruturais sobre milhares de proteínas pode ser obtido sob a forma de coordenadas cartesianas para cada átomo na proteína.
Muitos mais sequências de genes são conhecidos do que estruturas de proteínas. Além disso, o conjunto de estruturas resolvidas é inclinado para proteínas que podem ser facilmente submetidos às condições exigidas A cristalografia de raios-X, um dos principais métodos de determinação de estrutura. Em particular, as proteínas globulares são relativamente fáceis de cristalizar em preparação para a cristalografia de raios-X. Proteínas da membrana, pelo contrário, são difíceis de cristalizar e estão sub-representadas no APO. Iniciativas genômica estrutural tentaram remediar estas deficiências, resolvendo sistematicamente estruturas representativas dos principais classes de dobra. Proteína métodos de predição estrutura tenta proporcionar um meio de gerar uma estrutura plausível para proteínas cujas estruturas não foram experimentalmente determinados.
Funções celulares
As proteínas são os principais atores dentro da célula, disse estar a exercer as funções especificadas pela informação codificada em genes. Com exceção de certos tipos de RNA, a maioria das outras moléculas biológicas são elementos relativamente inertes sobre o qual as proteínas agem. Proteínas compõem metade do peso seco de um Células de Escherichia coli, ao passo que outras macromoléculas, tais como ADN e ARN constituem apenas 3% e 20%, respectivamente. O conjunto de proteínas expressas numa célula ou tipo de células em particular é conhecido como o seu proteoma.
A principal característica de proteínas que também permite que o seu conjunto diversificado de funções é a sua capacidade de se ligar outras moléculas especificamente e com força. A região da proteína responsável pela ligação de uma outra molécula é conhecida como a local de ligação e é muitas vezes uma depressão ou "bolso" na superfície molecular. Esta capacidade de ligação é mediada por a estrutura terciária da proteína, que define o local de ligação de bolso, e pelas propriedades químicas das cadeias laterais dos aminoácidos que rodeiam '. A ligação às proteínas pode ser extremamente apertado e específica; por exemplo, a proteína inibidora da ribonuclease humano liga-se a angiogenina com um sub-fentomolar constante de dissociação (<10 -15 M) mas não se liga de todo ao seu homólogo anfíbio ONCONASE (> 1 M). Alterações químicas extremamente pequenas, tais como a adição de um único grupo metilo para um parceiro de ligação pode, por vezes, suficiente para eliminar quase obrigatório; por exemplo, a aminoacil-ARNt-sintetase específico para o aminoácido valina discrimina a cadeia lateral muito semelhante do aminoácido isoleucina.
As proteínas se podem ligar a outras proteínas, bem como a substratos de pequenas moléculas. Quando as proteínas se ligam especificamente a outras cópias da mesma molécula, que pode oligomerizar para formar fibrilas; este processo ocorre frequentemente em proteínas estruturais, que consistem em monómeros globulares que a auto-associado para formar fibras rígidas. Interacções proteína-proteína, também regular a actividade enzima ti ca, por meio do controle da progressão ciclo celular, e permitir a montagem de grande complexos de proteínas que realizam muitas reações intimamente relacionados com uma função biológica comum. As proteínas também podem ligar-se a, ou até mesmo ser integrado no, membranas celulares. A capacidade dos produtos associados de ligação para induzir mudanças conformacionais em proteínas permite a construção de enormemente complexo redes de sinalização. É importante salientar, como interações entre proteínas são reversíveis, e dependem muito da disponibilidade de diferentes grupos de proteínas parceiras para formar agregados que são capazes de realizar conjuntos distintos de função, o estudo das interações entre proteínas específicas é a chave para entender aspectos importantes da a função celular, e, finalmente, as propriedades que distinguem tipos de células particulares.
Enzimas
O mais conhecido papel das proteínas na célula é tão enzimas que catalisam reacções químicas. As enzimas são normalmente altamente específica e acelerar apenas uma ou algumas reacções químicas. Enzimas efectuar a maior parte das reacções envolvidas metabolismo, assim como a manipulação de ADN em processos tais como a Replicação do DNA, reparo do DNA , e transcrição. Algumas enzimas actuam sobre outras proteínas para adicionar ou remover grupos químicos de um processo conhecido como modificação pós-translacional. Cerca de 4.000 reacções são conhecidas por ser catalisada por enzimas. A aceleração da velocidade conferida por catálise enzimática é frequentemente-enorme tanto quanto 10 17 vezes de aumento na taxa de reacção não catalisada através da no caso de descarboxilase orotate (78 milhões ano sem a enzima, 18 milissegundos com a enzima).
As moléculas ligadas e posta em prática por enzimas são chamados substratos. Ainda que as enzimas podem consistir em centenas de aminoácidos, normalmente é apenas uma pequena fracção dos resíduos que entram em contacto com o substrato, e uma fracção ainda menor e três a quatro resíduos de, em média, que estão directamente envolvidas na catálise. A região da enzima que se liga ao substrato e contém os resíduos catalíticos é conhecido como o sítio ativo.
A sinalização celular e a ligação do ligando
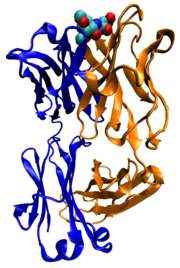
Muitas proteínas estão envolvidas no processo de sinalização celular e transdução de sinal. Algumas proteínas, como a insulina , são proteínas extracelulares que transmitem um sinal a partir da célula em que foram sintetizados para outras células na distante tecidos. Outros são proteínas da membrana que actuam como receptores cuja principal função é ligar uma molécula de sinalização e induzir uma resposta bioquímica na célula. Muitos receptores possuem um local de ligação exposto na superfície da célula e um domínio efector dentro da célula, o qual pode ter actividade enzimática ou podem ser submetidos a um alteração conformacional detectado por outras proteínas dentro da célula.
Os anticorpos são componentes proteicos de sistema imunológico adaptativo cuja principal função é a de vincular antigénios, ou substâncias estranhas no corpo, e orientá-las para destruição. Os anticorpos podem ser secretada para o meio extracelular ou ancorado nas membranas das especializado As células B conhecido como células plasmáticas. Considerando que as enzimas são limitados na sua afinidade de ligação para os seus substratos pela necessidade de realização de sua reação, os anticorpos não têm tais restrições. Afinidade de ligação de um anticorpo para o seu objectivo é extraordinariamente alta.
Muitas proteínas de transporte ligando se ligar nomeadamente pequenas biomoléculas e transportá-los para outros locais no corpo de um organismo multicelular. Estas proteínas devem ter uma elevada afinidade de ligação quando a sua ligando está presente em concentrações elevadas, mas também deve libertar o ligando quando está presente em concentrações baixas nos tecidos alvo. O exemplo clássico de uma proteína de ligação ao ligando é hemoglobina, que transporta oxigênio do pulmões para outros órgãos e tecidos em todos os vertebrados e tem perto homólogos em cada biológica reino. As lectinas são proteínas de ligação de açúcar que são altamente específicas para as suas porções de açúcar. As lectinas tipicamente desempenham um papel na biológica fenómenos de reconhecimento que envolvam células e proteínas. Receptores e hormonas são proteínas de ligação altamente específicos.
Proteínas transmembranares podem também servir como proteínas de transporte ligando a que alteram a permeabilidade da membrana celular para moléculas e íons pequenos. A membrana tem uma sozinho núcleo hidrofóbico através dos quais moléculas polares ou carregados não podem difusa. As proteínas da membrana conter canais internos que permitem que tais moléculas para entrar e sair da célula. Muitos proteínas de canais iônicos são especializados para selecionar apenas um ião particular; por exemplo, potássio e sódio canais de frequência para discriminar e apenas um dos dois iões.
Proteínas estruturais
Proteínas estruturais conferem rigidez e rigidez de componentes biológicos de outra forma de líquido. A maioria das proteínas são estruturais proteínas fibrosas; por exemplo, actina ea tubulina são globular e solúvel como monómeros, mas polimerizar para formar, fibras rígidas de comprimento que compreendem a citoesqueleto, que permite que a célula para manter a sua forma e tamanho. Colágeno e elastina são componentes críticos de tecido conjuntivo, tais como cartilagem, e queratina é encontrado em estruturas duras ou filamentosos tais como cabelo, unhas, penas , cascos, e alguns conchas de animais.
Outras proteínas que servem funções estruturais são proteínas motoras, tais como miosina, cinesina, e dineína, que são capazes de gerar forças mecânicas. Estas proteínas são cruciais para celular motilidade dos organismos unicelulares e do espermatozóides de muitos organismos multicelulares que reproduzem sexualmente. Elas também geram as forças exercidas por contratação músculos.
Métodos de estudo
Dado que algumas das moléculas biológicas mais habitualmente estudados, as actividades e as estruturas de proteínas são analisadas tanto in vitro e in vivo Estudos in vitro de proteínas purificadas em ambientes controlados são úteis para aprender como uma proteína leva a cabo a sua função:. por exemplo, cinética enzimática estudos explorar a mecanismo químico de actividade catalítica de uma enzima e a sua afinidade relativa para várias moléculas de substrato possíveis. Em contrapartida, em experimentos in vivo sobre as atividades das proteínas dentro das células ou até mesmo dentro de organismos inteiros podem fornecer informações complementares sobre onde uma proteína funciona e como ela é regulada.
Protein Purification
A fim de realizar em análises in vitro, uma proteína deve ser purificado a partir de outros componentes celulares. Este processo geralmente começa com lise celular, em que uma membrana de célula é interrompido e os seus conteúdos internos libertado para uma solução conhecida como um lisado bruto. A mistura resultante pode ser purificado utilizando ultracentrifugação, que fracciona os vários componentes celulares em fracções contendo proteínas solúveis; membrana de lipídios e proteínas; celular organelos, e ácidos nucleicos. Precipitação por um método conhecido como salga pode concentrar as proteínas a partir deste ligado. Vários tipos de cromatografia são então utilizados para isolar a proteína ou proteínas de interesse com base em propriedades, tais como peso molecular, carga líquida e afinidade de ligação. O nível de purificação pode ser monitorizado utilizando vários tipos de electroforese em gel se o peso molecular da proteína desejada e ponto isoeléctrico são conhecidos, por espectroscopia de se a proteína tem características espectroscópicas distinguíveis, ou pela Ensaios Enzimáticos se a proteína tem actividade enzimática. Além disso, as proteínas podem ser isoladas de acordo com a sua carga usando Eletrofocalização.
Para as proteínas naturais, uma série de passos de purificação podem ser necessários para obter proteína suficientemente pura para aplicações laboratoriais. Para simplificar este processo, engenharia genética é muitas vezes usado para adicionar recursos químicos para proteínas que tornam mais fácil para purificar sem afetar sua estrutura ou atividade. Aqui, uma "etiqueta" que consiste de um sequência de aminoácidos específica, muitas vezes de uma série de resíduos de histidina (um " His-tag "), está ligado a um terminal da proteína. Como resultado, quando o ligado foi passado por uma coluna de cromatografia contendo níquel , os resíduos de histidina ligar o níquel e anexar à coluna enquanto os componentes não marcados do lisado passe desimpedido. Um certo número de etiquetas diferentes foram desenvolvidos para ajudar os investigadores a purificar proteínas específicas a partir de misturas complexas.
Localização celular
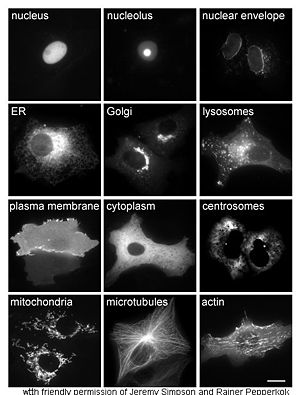
O estudo de proteínas in vivo é muitas vezes preocupados com a síntese e a localização da proteína dentro da célula. Embora muitas proteínas intracelulares sejam sintetizadas no citoplasma e membrana-limite ou proteínas secretadas no retículo endoplasmático, os detalhes de como as proteínas são direcionados para organelas específicas ou estruturas celulares sempre é claro. Uma técnica útil para avaliar a localização celular usa a engenharia genética para expressar de uma célula proteína de fusão ou quimera que consiste da proteína natural de interesse ligado a um " repórter ", tais como proteína verde fluorescente (GFP). A posição da proteína de fusão no interior da célula pode ser limpa e eficiente visualizada usando microscopia, como mostrado na figura oposto.
Outros métodos para elucidar a localização celular das proteínas requer a utilização de marcadores conhecidos compartimentados para regiões tais como o RE, o Golgi, lisossomas / vacúolos, mitocôndrias, cloroplastos, membrana de plasma, etc. Com o uso de fluorescente etiquetado versões desses marcadores ou de anticorpos para marcadores conhecidos, torna-se muito mais simples para identificar a localização de uma proteína de interesse. Por exemplo, imunofluorescência indireta permitirá a co-localização de fluorescência e demonstração de localização. Corantes fluorescentes são usados para etiquetar compartimentos celulares para uma finalidade semelhante.
Existem outras possibilidades, como bem. Por exemplo, imunohistoquímica geralmente utiliza um anticorpo para uma ou mais proteínas de interesse que estão conjugados com enzimas que produzam ou sinais cromogénicos ou luminescente que pode ser comparada entre as amostras, permitindo que a informação de localização. Outra técnica é aplicável cofractionation em sacarose (ou outro material) usando gradientes centrifugação isopícnica. Embora esta técnica não prova co-localização de um compartimento de densidade conhecida e a proteína de interesse, ele faz aumentar a probabilidade, e é mais susceptível de estudos em grande escala.
Finalmente, o método padrão de ouro-localização celular é imunomicroscopia. Esta técnica também utiliza um anticorpo para a proteína de interesse, juntamente com as técnicas clássicas de microscopia electrónica. A amostra é preparada para exame microscópico de electrões normal, e depois tratou-se com um anticorpo para a proteína de interesse que é conjugado com um material extremamente electro-denso, normalmente ouro. Isto permite a localização de ambos os detalhes ultra-estruturais, bem como a proteína de interesse.
Através de outro aplicativo conhecido como engenharia genética mutagénese dirigida, os pesquisadores podem alterar a sequência da proteína e, consequentemente, a sua estrutura, localização celular, e susceptibilidade à regulamentação. Esta técnica permite mesmo que a incorporação de aminoácidos não naturais em proteínas, utilizando ARNt modificados, e pode permitir o desenho racional de novas proteínas com propriedades novas.
Proteômica e bioinformática
O complemento total de proteínas presentes numa altura em um tipo de célula ou de células é conhecida como o seu proteoma, e o estudo de tais conjuntos de dados em grande escala define o campo de proteômica, nomeado por analogia ao campo relacionado de genómica. Técnicas experimentais chave em proteômica incluem Electroforese 2D, que permite a separação de um grande número de proteínas, espectrometria de massa , o que permite a identificação rápida de alto rendimento de proteínas e sequenciação de péptidos (na maioria das vezes, depois digestão em gel), microarrays da proteína, que permitem a detecção dos níveis relativos de um grande número de proteínas presentes em uma célula, e rastreio de dois híbridos, o que permite a exploração sistemática de interacções proteína-proteína. O complemento total de tais interacções biologicamente possíveis é conhecido como o interactome. A tentativa sistemática para determinar as estruturas das proteínas representando cada dobra possível é conhecido como genômica estrutural.
A grande quantidade de dados genómicos e proteomic disponíveis para uma variedade de organismos, incluindo o genoma humano, permite aos pesquisadores identificar de forma eficiente proteínas homólogas em organismos distantemente relacionadas por alinhamento de sequências . Ferramentas de sequências de perfil pode executar manipulações de sequências mais específicos, como mapas de enzimas de restrição, análises de leitura aberta para sequências de nucleótidos, e previsão de estrutura secundária. A partir destes dados árvores filogenéticas podem ser construídas e evolutivos hipóteses desenvolvido utilizando software especial como ClustalW sobre a ascendência dos organismos modernos e os genes que expressam. O campo da bioinformática busca de montar, anotar e analisar dados de genômica e proteômica, aplicando computacionais técnicas para problemas biológicos, tais como constatação de genes e cladística.
Previsão de estrutura e de simulação
Complementar ao campo da genômica estrutural, a previsão da estrutura de proteínas procura desenvolver maneiras eficientes para fornecer modelos plausíveis para proteínas cujas estruturas ainda não foram determinadas experimentalmente. O tipo mais bem-sucedida de previsão de estrutura, conhecida como modelação de homologia, baseia-se na existência de uma estrutura de "molde" com semelhança de sequência com a proteína a ser modelado; objetivo 'genômica estrutural é fornecer representação suficiente em estruturas resolvidas para modelar a maioria daqueles que permanecem. Embora a produção de modelos precisos continua a ser um desafio quando as estruturas do modelo apenas remotamente relacionados estão disponíveis, tem sido sugerido que o alinhamento da sequência é o gargalo nesse processo, como modelos bastante precisas podem ser produzidos, se um alinhamento de sequências "perfeita" é conhecida. Muitos métodos de previsão de estrutura têm servido para informar o campo emergente da engenharia de proteínas, em que novas dobras de proteínas já foram desenhados. Um problema computacional mais complexa é a previsão das interacções intermoleculares, tal como em acoplamento molecular e proteína-proteína predição interacção.
Os processos de dobramento de proteínas e ligação pode ser simulada usando essa técnica como mecânica molecular, em particular, dinâmica molecular e Monte Carlo , que cada vez mais se aproveitam da paralela e computação distribuída ( Folding @ projecto inicial; modelagem molecular na GPU). A dobragem de pequenos domínios alfa-helicoidal da proteína tais como o capacete vilina eo HIV proteína acessória foram simulados com sucesso in silico , e métodos híbridos que combinam dinâmica molecular padrão com mecânica quântica cálculos permitiram exploração dos estados eletrônicos de rodopsinas.
Nutrição
Mais microorganismos e plantas podem biossintetizar todos os 20 padrões aminoácidos , enquanto que os animais (incluindo seres humanos) tem de obter alguns dos aminoácidos dos dieta. aminoácidos que um organismo não pode sintetizar por sua própria são referidos como aminoácidos essenciais. Enzimas-chave que sintetizam certos aminoácidos não estão presentes em animais, tais como - aspartoquinase, que catalisa o primeiro passo na síntese de lisina, metionina, e treonina de aspartato. Se os aminoácidos estão presentes no ambiente, os microorganismos podem conservar a energia, tomando-se os aminoácidos de seus arredores e regular negativamente as suas vias biossintéticas.
Nos animais, os aminoácidos são obtidos através do consumo de alimentos que contêm proteínas. Proteínas ingeridas são, em seguida, dividido em aminoácidos através de digestão, o que tipicamente envolve a desnaturação da proteína através de exposição a ácido e a hidrólise por enzimas denominadas proteases. Alguns aminoácidos ingeridos são utilizados para a síntese proteica, ao passo que outros são convertidos em glicose através da gluconeogénese, ou introduzido a ciclo do ácido cítrico. Esta utilização de proteínas como combustível é particularmente importante sob condições de fome, uma vez que permite que as proteínas do próprio corpo para ser utilizado para suportar a vida, particularmente as encontradas em muscular. Aminoácidos são também uma importante fonte dietética de azoto .
História e etimologia
As proteínas foram reconhecidas como uma classe distinta de moléculas biológicas no século XVIII por Antoine Fourcroy e outros, distingue-se pela capacidade das moléculas para coagular ou flocular sob tratamentos com calor ou ácido. Exemplos observou na época incluía albumina dos ovos brancos, sangue albumina do soro, de fibrina, e trigo glúten. Químico holandês Gerhardus Johannes Mulder realizada análise elementar de proteínas comuns e descobriu que quase todas as proteínas tiveram a mesma fórmula empírica, C 400 H 620 N 100 O 120 P 1 S 1 . Ele chegou à conclusão errônea de que eles podem ser compostos por um único tipo de (muito grande) molécula. O termo "proteína" para descrever estas moléculas foi proposta em 1838 pelo associado de Mulder Jöns Jakob Berzelius; proteína é derivada do grego palavra πρωτεῖος ( proteios ), que significa "primário", "na liderança", ou "pé na frente". Mulder passou a identificar os produtos de degradação de proteínas, tais como o ácido amino leucina para a qual ele encontrou um peso molecular (quase correta) de 131 Da.
A dificuldade de purificação de proteínas em grandes quantidades fez muito difícil para os bioquímicos de proteínas primeiros a estudar. Assim, os estudos iniciais centraram-se em proteínas que podem ser purificadas em grandes quantidades, por exemplo, aqueles de sangue , clara de ovo, várias toxinas, e digestivos / enzimas metabólicos obtidos a partir de matadouros. Na década de 1950, o cão Co. Armour Hot purificado 1 kg de puro bovina pancreática ribonuclease A e disponibilizado gratuitamente para os cientistas; este gesto ajudou ribonuclease A tornar-se um dos principais alvos de estudo bioquímico para as décadas seguintes.
Linus Pauling é creditado com a previsão bem-sucedida de proteína regulares estruturas secundárias baseadas em ligações de hidrogênio, uma idéia primeiro apresentadas por William Astbury em 1933. Mais tarde trabalho por Walter Kauzmann sobre desnaturação, baseado em parte em estudos anteriores por Kaj Linderstrøm-Lang, contribuiu com um compreensão de dobragem e a estrutura da proteína mediadas por interacções hidrofóbicas. Em 1949 Fred Sanger correctamente determinada a sequência de aminoácidos da insulina , assim, demonstrar conclusivamente que as proteínas consistiu de polímeros lineares de aminoácidos, em vez de cadeias ramificadas, colóides, ou cyclols. As primeiras estruturas atômica resolução de proteínas foram resolvidas por cristalografia de raios-X na década de 1960 e por RMN na década de 1980. A partir de 2009, o Protein Data Bank tem mais de 55.000 estruturas atômica resolução de proteínas. Em tempos mais recentes, microscopia crio-eletrônica de grandes montagens macromoleculares e computacional previsão da estrutura de proteínas de pequenas proteínas domínios são dois métodos que se aproximam resolução atômica.